こんにちは。産婦人科医のtommyです。(Twitter:@obgyntommy)
この記事では、乳がんのデータセットを用いた教師あり機械学習の一通りの流れをscikit-learnを用いて学びます。
教師あり機械学習の一般的な流れは以下の通りです。
教師あり学習の機械学習の流れ
- データセットの読み込み
- データの前処理
- 探索的データ解析(EDA;Explanatory Data Analysis)
- 機械学習予測モデルの作成
- 性能評価
scikit-learnを用いた機械学習の流れについては、以下の記事を参照して下さい。
-

【機械学習】scikit-learnの使い方【基礎から全て解説】
続きを見る
又、Google Colaboratoryの使い方については以下の記事を参照して下さい。
-

Google Colaboratoryの使い方【完全マニュアル】
続きを見る
尚、ワインのデータセットを用いた教師ありの機械学習についてはこちらの記事をご参照下さい。
-

【教師あり学習】機械学習でワインの品質判定を行ってみよう【scikit-learn】
続きを見る
Google Colaboratoeyを使用して学習される方は、こちらのリンクを参照して下さい。
では早速学習していきましょう。
Breast cancerデータセットの読み込みと内容確認

skleanのライブラリから「breast cancer」のデータセットを読み込みます。
breast cancerのデータセットは乳がん患者から採取した細胞の情報(半径や滑らかさ)から悪性なのか良性なのかを判断するためのデータセットになります。
breast cancerデータセットの中身を確認します。
In[]
1 2 3 | from sklearn.datasets import load_breast_cancer data_breast_cancer = load_breast_cancer() data_breast_cancer |
Out[]

※ 全ての結果は反映できていません。
このデータセットの中身はPythonの辞書型になっていますので、取得したい対象のキーを以下のように指定することにより対象の中身(バリュー)を取得できます。
以下のコードでは正解ラベルの取得を行なっています。
In[]
1 | data_breast_cancer["target"] |
Out[]

データの前処理

次に $X$ を特徴量、$y$ を正解ラベルとし前処理を行なっていきます。まずは正解ラベルの前処理を行います。
元のデータは0, 1という整数型のデータになっていますが、今回は分類問題のためにこれをカテゴリカル変数に変換します。
target_namesキーにカテゴリ名があるのでこれを使いましょう。
- malignant:悪性
- benign:良性となります。
カテゴリカル変数とは
変数には「質的変数」「量的変数」があります。
質的変数は「カテゴリー変数(categorical variable、カテゴリカル変数)」と呼ばれる事もあります。
In[]
1 2 3 4 | import pandas as pd y_all = pd.DataFrame(data_breast_cancer["target"],columns=["target"]) y_all = y_all.replace({0:data_breast_cancer["target_names"][0], 1:data_breast_cancer["target_names"][1]}) y_all.head() |
Out[]

続いて、特徴量の前処理を行います。特徴量の名前は feature_names 、値は dataキー に含まれていますのでそれを用います。
In[]
1 2 | X_all = pd.DataFrame(data_breast_cancer["data"],columns=data_breast_cancer["feature_names"]) X_all.head() |
Out[]

※ 一部のみを記載しています。
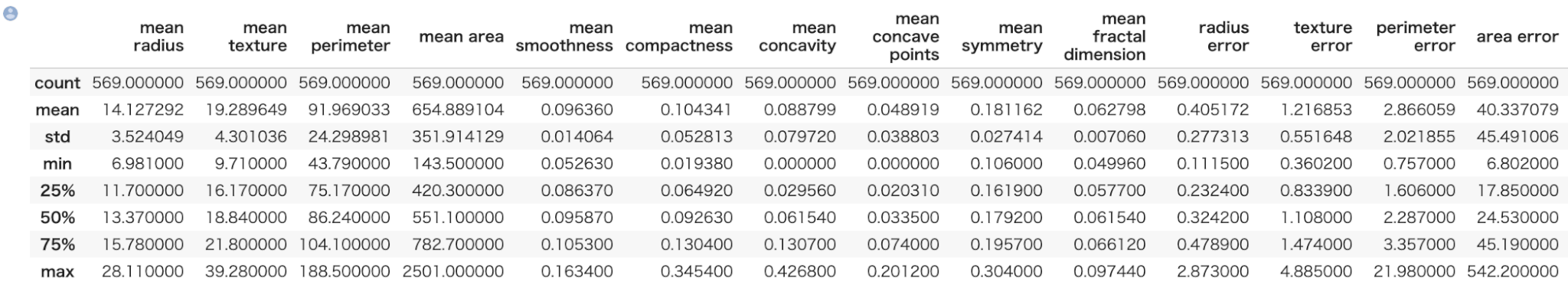
describeメソッドによって、一括で全ての特徴量の統計値の概要を表示できます。
In[]
1 | X_all.describe() |
Out[]
続いて、全てのデータを学習用と評価用に分割します。これには sklearn の train_test_split メソッドを使います。
学習用データと評価用データの数の割合ですが、今回は 2:1 とします。
※ 2:1でなければならないというわけではなく、一般的には評価用データ数が全体の2-4割程度にすることが多いです。
In[]
1 2 | from sklearn.model_selection import train_test_split X_train, X_test, y_train, y_test = train_test_split(X_all, y_all, test_size=0.33, random_state=0) |
学習データの特徴量と正解ラベルを1つのデータセットとしてまとめます。
In[]
1 | train = pd.concat([X_train,y_train],axis=1,sort=False) |
続いて、学習用データセットの特徴量と正解ラベルの型を確認します。pandas の info メソッドにより全てのカラムの型を確認できます。
pandasの使い方は以下の記事で基本から応用までを学習することが出来ますので、参照してください。
In[]
1 | train.info() |
Out[]

全ての特徴量が浮動小数点数(float型)ということが確認できました。
探索的データ解析(EDA)

続いて、このデータに関して探索的データ解析(EDA)を行なっていきます。
探索的データ解析(EDA)の目的ですが、これから機械学習を使って分類を行なっていきますが、その前に『分類が可能そうかそうでないか』を見極めるのが重要となります。
※ 目的なくデータ全体の平均や分散を算出するだけでは意味がありません。
このデータセットは特徴量が浮動小数点数(float型)で構成されているため、値の大小でクラス分けが大体できれば理想的になります。
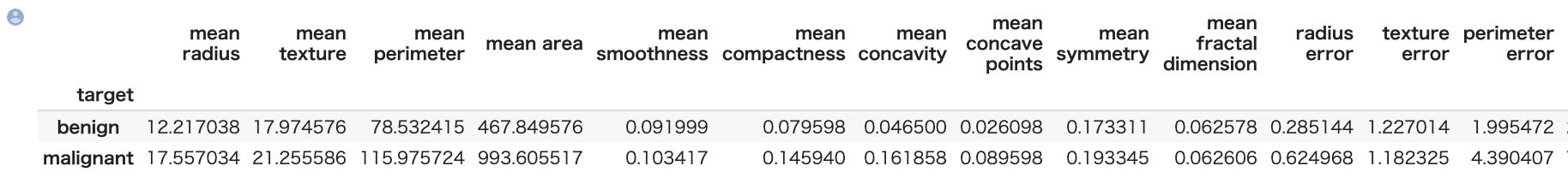
クラス、特徴量ごとの平均をまずは算出してみます。
In[]
1 | train.groupby("target").mean() |
Out[]
※ 一部のみを表示しています。
全体的に中央全体傾向としてどの変数も malignant > benign となっています。
また、" mean concavity", "area error", "worst area"あたりの特徴量が良性悪性間に差があることが分かります。
In[]
1 | train.groupby("target").mean()[["mean concavity","area error","worst area"]] |
Out[]

続いてこれらの特徴量をもう少し詳細に確認していきます。
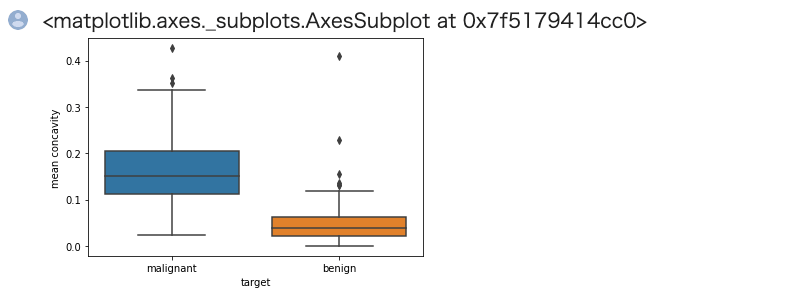
箱ひげ図を用いることによりデータの分布を考慮した良性悪性間の違いを確認します。 描画には seaborn ライブラリの boxplot メソッドを使うと便利です。
In[]
1 2 | import seaborn as sns sns.boxplot(x='target', y='mean concavity', data=train) |
Out[]
In[]
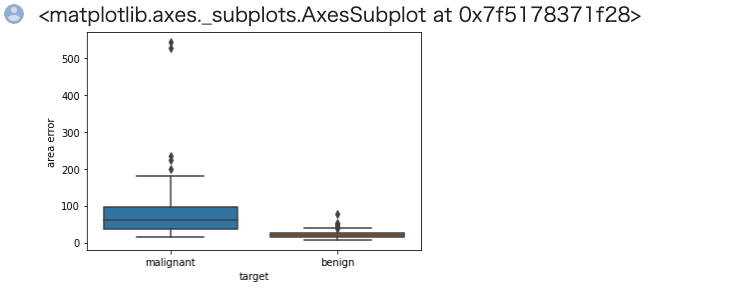
1 2 | import seaborn as sns sns.boxplot(x='target', y='area error', data=train) |
Out[]
In[]
1 2 | import seaborn as sns sns.boxplot(x='target', y='worst area', data=train) |
Out[]
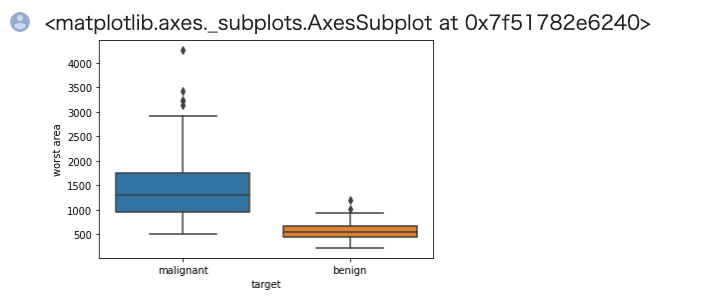
若干の外れ値はありますが、25%-75%点の部分で良性悪性間の被りが無いので分類はたやすそうです。
続いて散布図により、2つの特徴量で良性悪性が上手く分類できそうかを確認してみます。 これも seaborn の scatterplot メソッドを使うと便利です。
Pythonのseabornの使い方については以下の2つの記事を参照して復習して下さい。
-

PythonのライブラリSeabornの使い方【前編】
続きを見る
-

PythonのライブラリSeabornの使い方【後編】
続きを見る
In[]
1 | sns.scatterplot( x='mean concavity', y='worst area',hue="target", data=train) |
Out[]
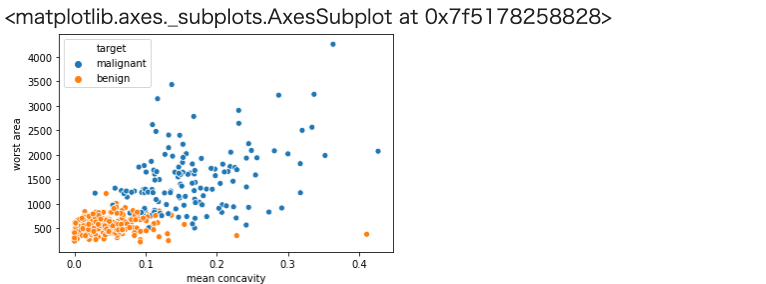
In[]
1 | sns.scatterplot( x='area error', y='worst area',hue="target", data=train) |
Out[]
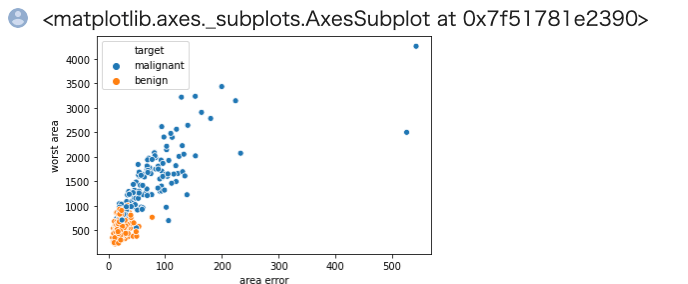
以上から、良性悪性が上手く纏まっており、機械学習などの手法により分類が可能そうという目処がつきました。
よってこの結果より、分類を行うモデルの作成を行なっていきます。
機械学習予測モデルの作成

これから機械学習予測モデルの作成を行います。
すでに前処理は完了していますので、機械学習モデルのハイパーパラメータ探索から行います。
今回は機械学習モデルとしてランダムフォレスト、ハイパーパラメータ探索としてグリッドサーチを用います。
両方ともsklearnのクラスとして用意されています。
In[]
1 2 | from sklearn.ensemble import RandomForestClassifier from sklearn.model_selection import GridSearchCV |
ランダムフォレストのハイパーパラメータについては色々ありますが、今回は代表的なもののみ取り上げることにします。
max_depth(木深さ)n_estimators(木の数)min_samples_split(木ノードにおいてそれ以上分割を行うための最少サンプル数)
GridSearchCVの引数について解説します。
scoring については今回は accuracy にしております。
※ ただし不均衡データの場合は accuracy からは変えたほうが良い場合があります。これは別途解説します
CVはクロスバリデーションにおけるデータセットの分割数ですが、これは一般的には3-10くらいの場合が多いです。
もちろん分割数が多いほど結果の信頼性が高まりますが、その分計算時間が増大します。
グリッドサーチの手順としては、ハイパーパラメータの候補を作成し、それを元にグリッドサーチのインスタンスを作成、データをフィッティングさせます。
グリッドサーチの手順まとめ
- ハイパーパラメータの候補を作成
- グリッドサーチのインスタンスを作成
- データをフィッティングさせる
In[]
1 2 3 4 5 6 7 8 9 | param_grid = {"max_depth":[1,2,3,5,7], "n_estimators":[100,200,500],"min_samples_split":[2,3, 5,7] } clf = GridSearchCV(estimator=RandomForestClassifier(random_state=0), param_grid = param_grid, scoring="accuracy", cv = 5, n_jobs = -1) clf.fit(X_train,y_train["target"].values) |
Out[]

どのハイパーパラメータが最も良い性能を及ぼすかは、フィッティングさせたインスタンス(cld f)の best_params_ メソッドで確認することができます。
In[]
1 | print("Best Model Parameter: ",clf.best_params_) |
また、このハイパーパラメータを採用した際のモデルはbest_estimator_メソッドにより作成が可能です。
In[]
1 | clf_best = clf.best_estimator_ #best estimator |
性能評価

続いて、予測と性能評価を行いましょう。
※ 性能は正解データと予測データがどの程度合致しているかということですが、一般的にはprecision、recall、f1-score、accuracyの4種類があります。(説明は割愛しますが、本来はデータ分析の背景からこのどれを選ぶかを決定することが多いです。)
今回は元々性能指標として設定していたaccuracyに注目することにしましょう。
accuracy含め、性能評価結果は sklearn の classification_report によって一括で算出することが可能です。
In[]
1 2 3 | from sklearn.metrics import classification_report y_true, y_pred = y_test, clf_best.predict(X_test) print(classification_report(y_true, y_pred)) |
Out[]
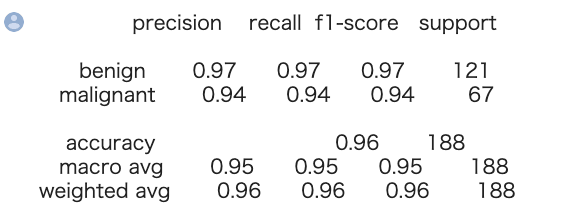
以上の結果から、十分な分類性能が得られていることが分かります。
